此项研究主要受到国家重点研发计划项目“高致病性病毒转录复制过程关键蛋白质机器的功能和干预机制”(2018YFA0507200,项目首席科学家为中科院武汉病毒所陈新文研究员)的支持。论文的四位共同第一作者中:博士生王美华、舒波(已获博士学位)主要完成了前两类转位中间体的工作;博士生李瑞主要完成了基序G突变体的酶学表征并与王美华共同完成了利用基序G突变体获得第三类转位中间体的工作;博士后景旭平主要参与了RdRP-RNA复合物晶体衍射数据收集和结构解析工作。叶寒青副研究员参与了基序G突变体的酶学表征工作,龚鹏研究员为论文通讯作者。相关论文2020年5月25日以“Stringent control of the RNA-dependent RNA polymerase translocation revealed by multiple intermediate structures”(多种中间体结构揭示RdRP对转位过程的严格控制)为题在Nature Communications上在线发表。
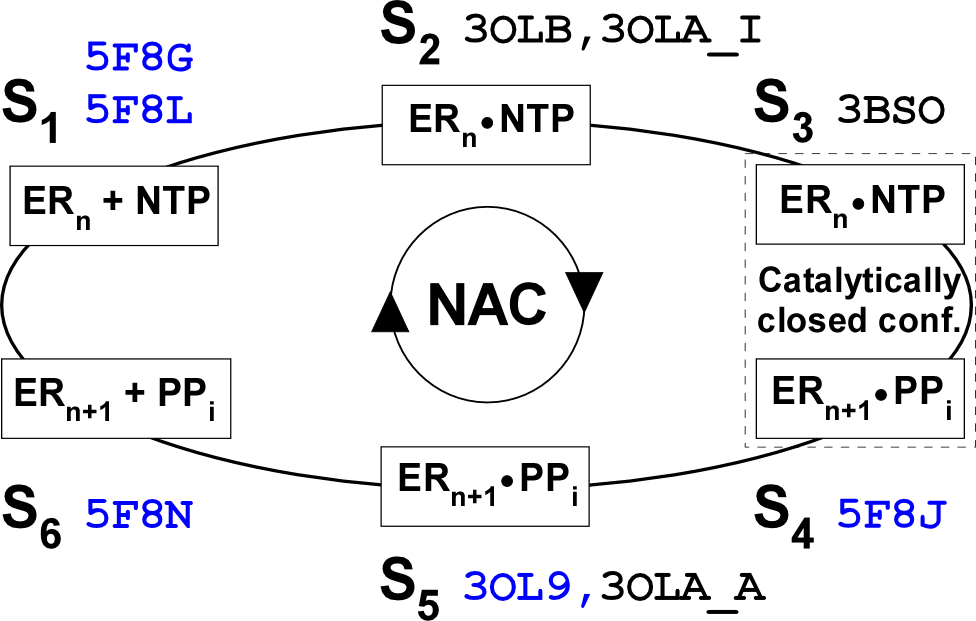
图1. 病毒RdRP的六状态核苷酸添加循环模型。核苷酸添加循环(nucleotide addition cycle或NAC)可由六种状态(S1-S6)阐明:S1 - RdRP催化中心未结合NTP,为开放式构象;S2 - NTP进入催化中心,仍为开放式构象;S3 - 催化中心关闭;S4 - 化学反应发生后,RNA产物链延伸一个核苷酸,仍保持关闭式构象;S5 - 催化中心转换为开放式构象;S6 - 转位中间体。图中列出了每种状态对应的结构PDB号(四位代码)以及用于判断转位中间体状态的参考PDB号(蓝色字体)。
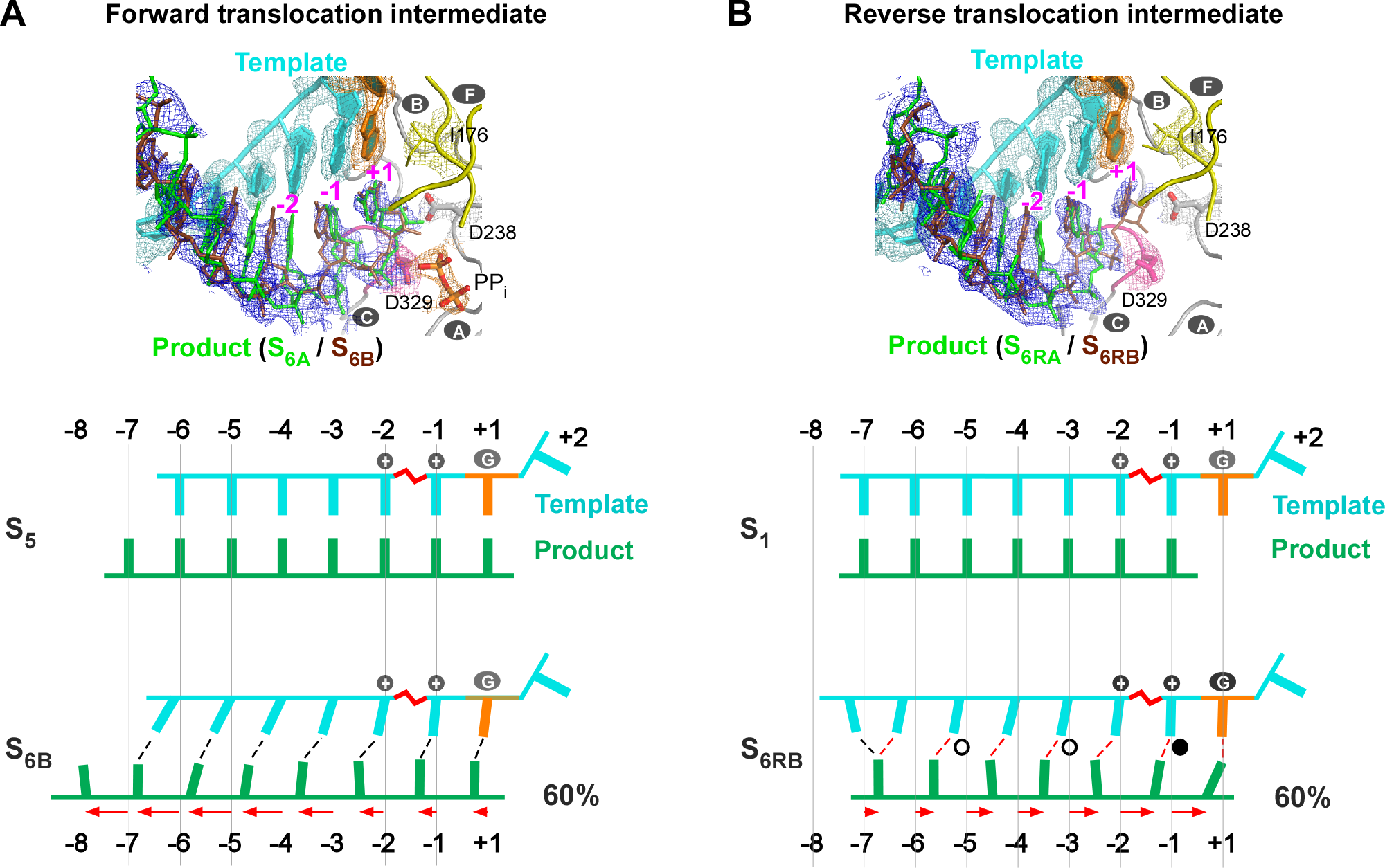
图2. 新解析的RdRP正向与逆向转位中间体结构。在每种转位中间体结构的产物链(product)均由两种混合态(正向:S6A/S6B;逆向:S6RA/S6RB)构成,展示了产物链先于模板链运动的特征,其中正向转位(A)RNA双链碱基对得以维持(黑色虚线),而逆向转位双链间发生滑动(红色虚线显示新形成的碱基对,其中实心和空心圆标识分别表示常规和摇摆碱基对)。
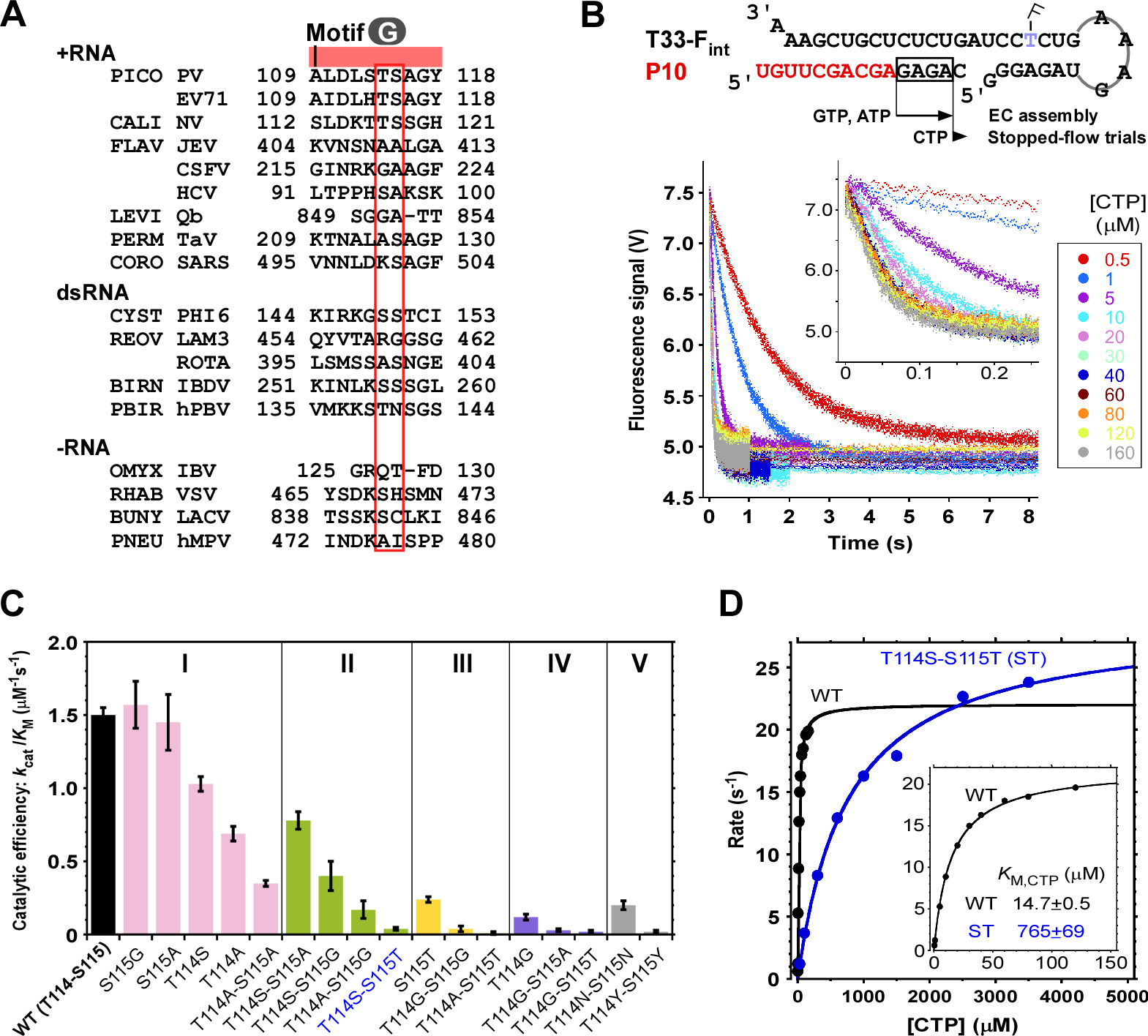
图3. 针对RdRP独特基序中参与模板链“锁定”的两个氨基酸位点的系统性酶学表征。A)RdRP在相关位点(红框标识)多为G/A/S/T等小侧链氨基酸。B)基于快速反应装置和荧光实时检测的RdRP单步反应表征方法。C)部分“锁定”位点突变体具有与野生型(WT)相近的催化效率(kcat/KM)。基于酶反应动力学特征将WT及突变体划分为五种不同类型(I-V)。D)T114S-S115T(ST)突变体KM值约为WT的50倍。
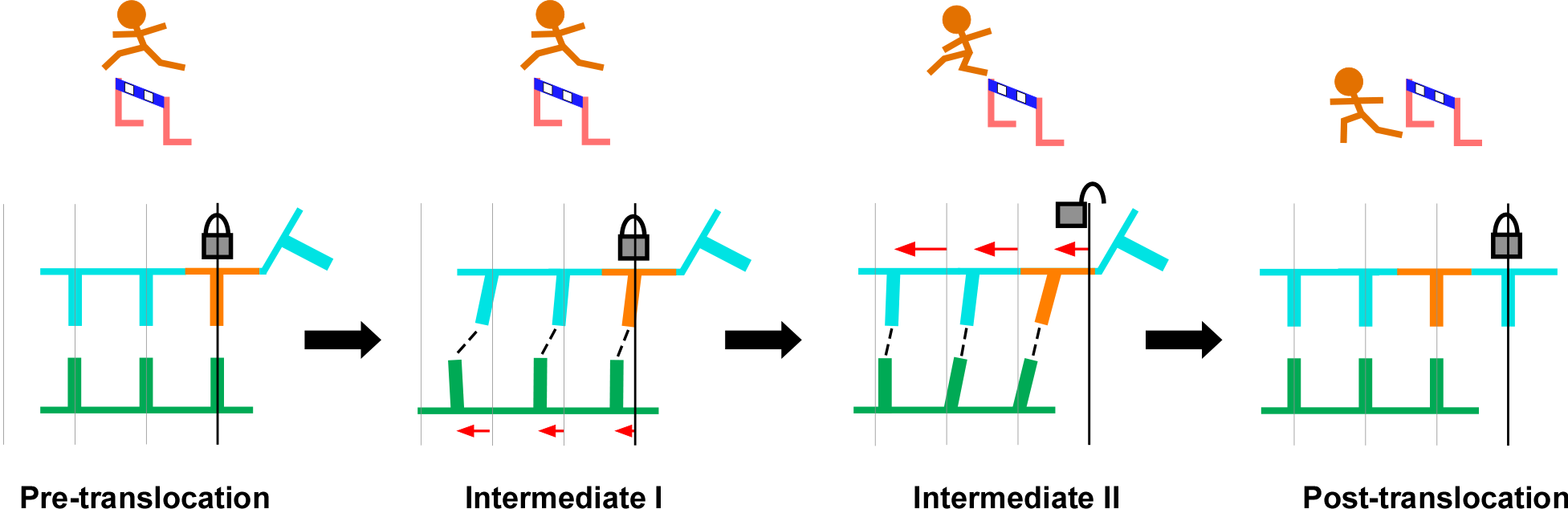
图4. RdRP分步不对称转位过程分解示意图。左:转位前,基序G锁定RNA模板链,对模板链来说,基序G则相当于跨栏运动员需要跨越的栏架;中一:在转位前期,产物链先于模板链发生移动,模板链仍处于锁定状态;中二:转位后期,锁定相互作用被打破,模板链发生移动,跨越基序G;右:转位后,模板+1位核苷酸(橘黄色)到达-1位,基序G重新锁定模板。
 附件下载:
附件下载:
科学研究